اولین ابزار رایگان بیوانفورماتیک ایران منتشر شد …

زمان تخمینی مطالعه: 14 دقیقه
متیلاسیون DNA نوعی نشانهگذاری شیمیایی است که در آن گروههای عاملی متیل (CH3) توسط آنزیمهایی با خاصیت متیل ترنسفرازی (انتقالدهنده گروه متیل) به DNA اضافه میشوند. این مکانیسم در ژنوم بسیاری از موجودات اتفاق میافتد و الگوی این نشانهگذاری بهصورت ارثی به نسل بعد منتقل میشود.
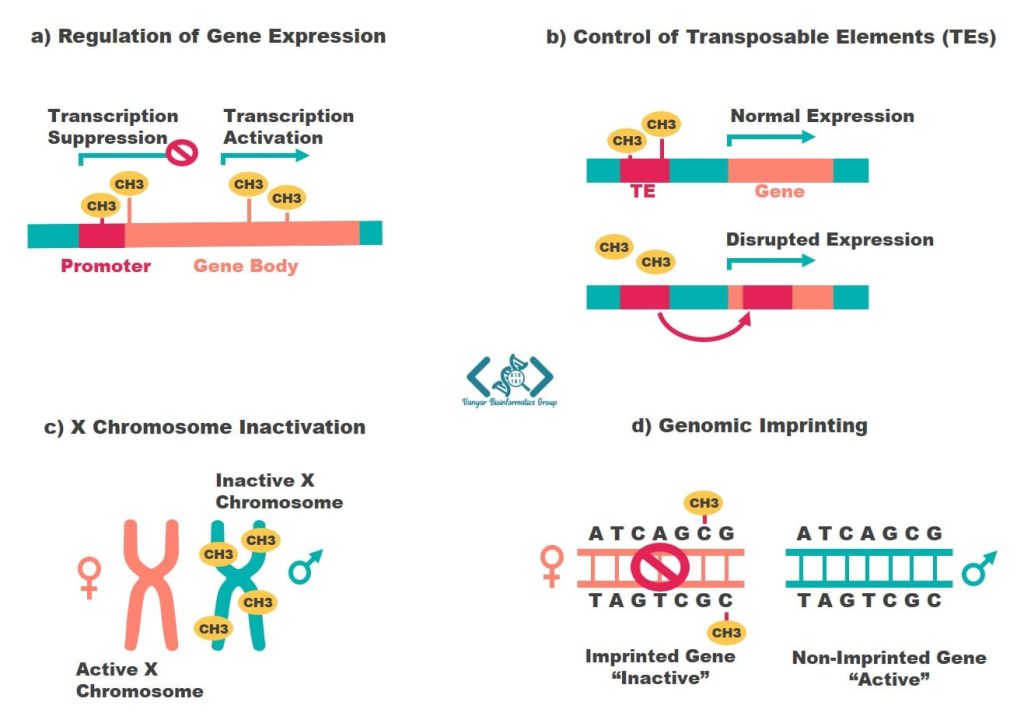
متیلاسیون DNA در نواحی مختلف ژنوم میتواند براساس توالی ژنتیکی آن ناحیه، تأثیرات متفاوتی بر فعالیت ژن داشتهباشد. بهطور کلی متیلاسیون در ۳ ناحیه از ژنوم انسان اتفاق میافتد:
نواحی بین ژنی (Intergenic)، جزایر CpG، و ژنبادی
متیلاسیون DNA در مناطق بین ژنی، عناصر قابل جابهجایی (Transposable) و ویروسی را که میتوانند در ژنوم حرکت کرده و باعث ایجاد اختلال در بیان ژن شوند یا برای ژنوم مضر باشند، خاموش میکند.
جزایر CpG درواقع نواحی از ژنوم هستند که چگالی CpG (نوکلئوتید C که نوکلئوتید G با فسفات به آن متصل است – ساختار DNA) بالایی دارند. این جزایر اغلب در پروموترها قرار دارند و در طول تکامل حفظ شدهاند. متیلاسیون DNA بهطور مستقیم از اتصال فاکتورهای رونویسی حساس به متیلاسیون، به نواحی اتصالی شان در پروموتر ژنها، ممانعت به عمل میآورد. در این مکانیسم باید دی نوکلئوتیدهای CpG در جایگاههای اتصال این فاکتورهای رونویسی حساسی به متیلاسیون قرار داشتهباشند.
پیشنهاد: دوره جامع طراحی پرایمر یک فرصت طلایی برای یادگیری حرفهای طراحی پرایمر در تکنیک DNA Methylation با استفاده از ابزارهای پرکاربرد در این حوزه است.

ژنبادی درواقع ناحیه رونویسیشونده یک ژن است (از نقطه آغاز رونویسی تا پایان رونویسی) (شامل اگزون / اینترون، و فاقد عناصر تنظیمی مانند پروموتر)، یعنی بخشی از ژن که به mRNA رونویسی میشود. براساس یافتهها متیلاسیون DNA در این ناحیه با بیان بالاتر ژن در سلولهای در حال تقسیم همراه است. این در حالی است که تأثیر متیلاسیون در اگزون اول ژن و همینطور پروموتر، با ژنبادی متفاوت است چرا که متیلاسیون این نواحی اغلب سبب خاموشی ژن میشود.
متیلاسیون DNA علاوهبر برای تنظیم بیان ژنها در انواع بافتهای بدن، در تکوین طبیعی نیز نقش بسیار مهمی در فرایندهایی کلیدی از جمله نقشگذاری ژنومی (Genomic Imprinting) و غیرفعال سازی کروموزوم X ایفا میکند.
متیلاسیون در موجودات پروکاریوتی مانند E. coli با عنوان متیلاسیون Dam و Dcm شناخته میشود. در نوع Dam، متیلازِ Dam گروه متیل را به آدنین، و در نوع Dcm، متیلترنسفرازِ Dcm، گروه متیل را به سیتوزین اضافه میکند.
متیلاسیون در موجودات یوکاریوتی اکثراً از نوع CpG است. در گیاهان، سیتوزینها میتوانند بهصورت متقارن (CG یا CHG) یا نامتقارن (CHH، که H میتواند A، T یا C باشد) در توالی ژنوم یافت شوند. در پستانداران، متیلاسیون DNA میتواند روی هر سیتوزینی از توالی رخ دهد، با این حال بیش از 98 درصد متیلاسیون DNA در دینوکلئوتیدهای CpG در سلولهای سوماتیک اتفاق میافتد.
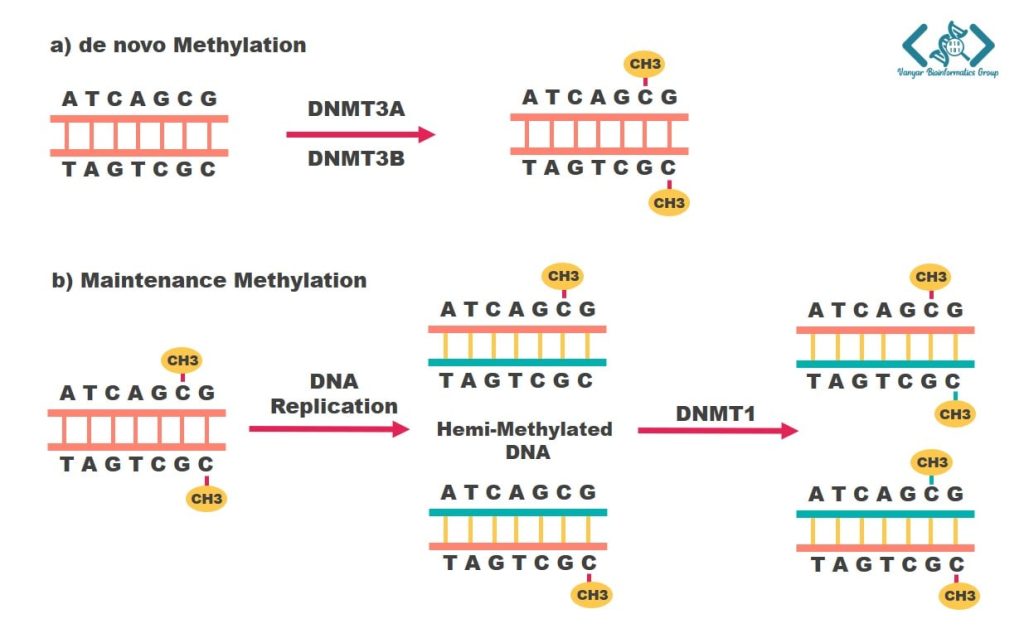
متیلاسیون DNA در اکثر موجودات یوکاریوتی بهواسطه آنزیمهای DNMT انجام میشود.
DNMT1: در طول همانندسازی DNA عمل میکند تا الگوی متیلاسیون را از رشته DNA والدین بر روی رشته دختر تازه سنتز شده کپی کند.
DNMT3A و DNMT3B: برخلاف DNMT1 میتوانند یک الگوی متیلاسیون جدید برای DNA فاقد الگوی متیلاسیون ایجاد کنند و بنابراین بهعنوان de novo DNMT شناخته میشوند.
بسیاری از مطالعات انسانی نشان میدهند که متیلاسیون نابجای DNA (شامل کاهش متیلاسیون یا هایپومتیلاسیون و افزایش متیلاسیون یا هایپرمتیلاسیون)، موجب القای بیماریزایی در بسیاری از بیماریها مانند انواع سرطانها، بیماریهای خودایمنی (مانند MS و آرتریت روماتوئید)، بیماریهای متابولیک (مانند دیابت و چاقی) و بیماریهای نورولوژیک (شامل اختلال طیف اوتیسم و بیماری پارکینسون) میشود. بر این اساس، تیمهای تحقیقاتی مختلفی به بررسی وضعیت متیلاسیون DNA مرتبط با پاتوژنز هر بیماری تمرکز کردهاند، زیرا ممکن است بهعنوان نشانگرهای زیستی مفیدی برای تشخیص، پیشآگهی، پایش بیماری یا پیشبینی پاسخ به درمان عمل کنند.
انتخاب تکنیکهای مناسب برای بررسی الگوی متیلاسیون DNA، نیازمند اشراف به هدف مطالعه است. براساس اینکه بخواهید (۱) تغییرات ناشناخته متیلاسیون را کشف کنید، یا (۲) متیلاسیون را در نواحی تنظیمی موردنظر یا ژنهای خاصی بررسی کنید، به تکنیک مختص خود نیاز دارید.
وقتی هدف شما کشف تغییرات ناشناخته متیلاسیون در ژنوم باشد، باید از تکنیکهایی استفادهکنید که به آنالیز الگوی متیلاسیون کل ژنوم میپردازند یا نواحی با الگوی متیلاسیون متفاوت (Differentially Methylated) را پیدا میکنند. این بحث در قسمت اپیژنوم مطرح میشود.
در این مقاله ما فرض میکنیم که میخواهید متیلاسیون را در نواحی تنظیمی یا ژنهای خاصی بررسی کنید، یا اینکه میخواهید الگوی متیلاسیون ناحیهای خاص که از روشهای با بازده بالا (High-throughput) بهدستآمدهاند را تایید کنید. به این منظور، از روشهایی استفاده میشود که اساس همه آنها را میتوانیم مطابق شکل زیر به سه دسته تقسیم کنیم. در ادامه مقاله هر دسته را شرح دادهایم.
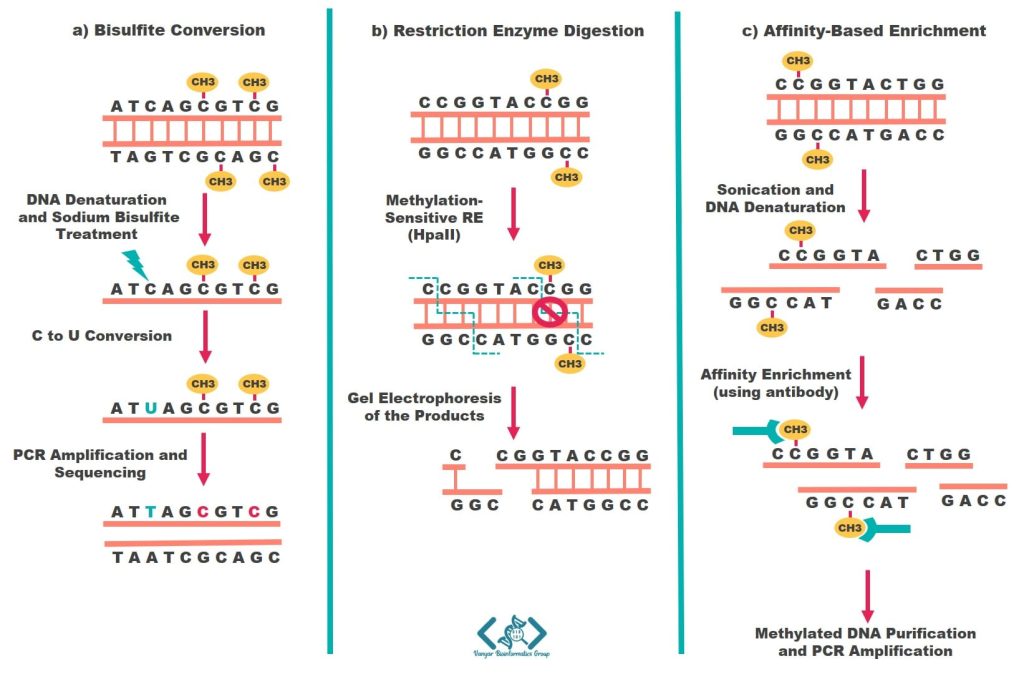
تبدیل بیسولفیت پرکاربردترین تکنیک برای بررسی الگوی متیلاسیون است و از بیسولفیت سدیم استفاده میکند. بیسولفیت سدیم با DNA واکنش میدهد و سیتوزینهای غیرمتیله را به یوراسیل تبدیل میکند، درحالیکه سیتوزینهای متیله را بدون تغییر باقی میگذارد. سپس این تبدیل را میتوان با استفاده از روشی مثل توالییابی Sanger تشخیص داد. با مقایسه توالی DNA غیر تغییریافته (قبل از اثر دهی با بیسولفیت سدیم) با توالی DNA تغییریافته (بعد از اثر دهی با بیسولفیت سدیم)، میتوانید جایگاه سیتوزینهای متلیه را شناسایی کنید. از جمله مزایای این روش میتوان به قابلیت آنالیز کمّی متیلاسیون تقریباً در هر نقطهای از ژنوم، و بررسی دقیق متیلاسیون CpG با وضوح تکنوکلئوتید اشاره کرد. معایب این روش هم میتواند شامل تجزیه DNA بهخاطر اثر بیسولفیت سدیم و چالشهای بیوانفورماتیکی طراحی پرایمر باشد.
MSP پرکاربردترین استراتژی آنالیز متیلاسیون DNA براساس تبدیل بیسولفیت است که همواره در تشخیص نمونههای بالینی به کار گرفتهمیشود. در این روش، DNA تیمار شده با بیسولفیت، بهعنوان الگوی PCR عمل میکند. MSP از دو مجموعه پرایمر استفاده میکند: پرایمرهای مخصوص CpG متیله و پرایمرهای مخصوص CpG غیرمتیله.
تاکنون فرمهای مختلفی از تکنیک MSP ابداعشده که همگی حساسیت بالایی در تشخیص دارند. تعدادی از این تکنیکها عبارتند از:
روشهای مبتنی بر آنزیم محدودکننده، از آنزیمهای حساس به متیلاسیون (Methylation-sensitive) و آنزیمهای وابسته به متیلاسیون (Methylation-dependent) استفاده میکنند که بهترتیب فقط DNA غیرمتیله و متیله را در جایگاه برش اختصاصی میبُرند. روش MS-AP-PCR (استفاده از آنزیمهای MspI یا HpaII) و AIMS (استفاده از آنزیمهای SmaI و XmaI) بر اساس حساسیت به متیلاسیون ابداع شدهاند. با این حال، هر دو تکنیک از رزولوشن و بازده پایین رنج میبرند، به کیفیت و مقدار DNA بالایی احتیاج داشته و از مواد رادیواکتیو استفاده میکنند. در نتیجه، امروزه بهندرت از اینها برای بررسی وضعیت متیلاسیون استفاده میشود.
تکنیک MeDIP یا رسوبگذاری ایمنی برای DNA متیله، از معروفترین تکنیکهای این دسته است.
در این روش DNA ژنومی را با سونیکاسیون قطعهقطعه میکنند. سپس قطعات واسرشته میشوند و با یک یا چند آنتیبادی شناساییکننده ۵-متیل-سیتوزین اثر دادهمیشوند. DNA متیله که اکنون به آنتیبادی متصل است را خالصسازی کرده و از آن برای تکثیر با PCR استفاده میکنند.
روشهای مبتنی بر تمایل، امکان ارزیابی سریع و راحت تغییرات متیلاسیون را فراهم میکنند و بهصورت تجاری در دسترس هستند. با اینحال، این روشها به میزان بالایی از DNA نیاز دارند، بهدلیل اتصالات غیراختصاصی به DNA غیرمتیله، دارای پتانسیل نتایج مثبت کاذب هستند و اطلاعات کمّی بهدست نمیدهند.
*** علاوهبر این سه استراتژی مطرحشده، تکنیکهایی هم وجود دارند که ترکیبی از این اصول هستند. مانند تکنیک COBRA که آمیزهای از تبدیل بیسولفیت و هضم با آنزیم محدودکننده میباشد.
بهطور کلی، برای طراحی پرایمرهای مخصوص آنالیز متیلاسیون، سه استراتژی بسته به هدف و روش مطالعه پیشروی شما وجود دارد:
این پرایمرها بین توالی هدف در صورتی که متیله باشد یا غیرمتیله، تفاوت قائل میشوند. به این شکل که باید برای مناطقی طراحی شوند که CpGهای موردمطالعه شما در آن قرار دارند. در صورتی که این CpG ها متیله باشند، پرایمرهایی که برای CpGهای متیله طراحی میکنید، به الگو وصل میشوند. اما در صورتی که این CpGها غیرمتیله باشند، تنها پرایمرهایی که برای هدفگیری CpG غیرمتیله بهینه کردهاید، میتوانند توالی را شناسایی کرده و به آن متصل شوند.
اگر ناحیه موردنظر شما با پرایمر مخصوص CpGهای متیله تکثیر شود، نشان از آن دارد که CpG در آن ناحیه متیله است ولی اگر با پرایمر مخصوص CpGهای غیرمتیله تکثیر شود، بیانگر آن است که متیلاسیونی در محل شناسایی وجود ندارد. درنهایت، وضعیت متیلاسیون CpGهای هدف را میتوانید با تصویربرداری ژل الکتروفورز تعیین کنید.
پرایمرهای اختصاصی متیلاسیون باید حداقل یک محل CpG در انتهای ‘3 خود و تعداد کافی سیتوزین غیرCpG در طول توالی داشتهباشند. سیتوزینهای غیرCpG غیرمتیله اند و طی تبدیل بیسولفیت به یوراسیل تبدیل میشوند. وجود این سیتوزینها در توالی پرایمر موجب میشود تا فقط DNA تغییریافته با بی سولفیت تکثیر شود و DNA تغییر نیافته، تکثیر نشود.
پیشنهاد: افزایش فرصتهای شغلی و تحقیقاتی با شرکت در دوره بیوانفورماتیک عمومی و کسب مهارتهای اساسی بیوانفورماتیک.

این پرایمرها باید در مناطقی واقع شوند که CpG وجود ندارد تا ناحیه هدف (حاوی چندین CpG) را صرفنظر از متیله و غیرمتیله بودن، تکثیر کنند. در این استراتژی نیز سیتوزینهای غیرCpG در توالی پرایمرها وجود دارند تا به افتراق DNA تغییریافته با بیسولفیت از DNA تغییرنیافته، کمک کنند. بعد از انجام PCR، بایستی محصول را توالییابی کنید تا بتوانید وضعیت CpGهای توالی هدف را مشخص کنید.
پیشنهاد: بهمنظور ثبت سفارش طراحی پرایمر برای بررسی متیلاسیون DNA به صفحه تهیه پلنهای طراحی پرایمر و پروب مراجعه فرمایید. طراحی پرایمر در میزکار وانیار و زیرنظر کارشناسان بیوانفورماتیک صورت میگیرد.
این پرایمرها برای تکثیر ناحیه موردنظر که حاوی CpG قابل شناسایی برای آنزیم محدودکننده است، طراحی میشوند. این پرایمرها به اثر دهی سدیم بیسولفیت نیاز ندارند بلکه بر هضم افتراقی DNA متیله و غیرمتیله توسط آنزیم محدودکننده تکیه دارند. پرایمرها باید محل شناسایی آنزیم محدودکننده را در محصول PCR داشته باشند، اما نه در محل اتصال پرایمر.
Moore LD, Le T, Fan G. DNA methylation and its basic function. Neuropsychopharmacology. 2013 Jan;38(1):23-38.
Jin Z, Liu Y. DNA methylation in human diseases. Genes & diseases. 2018 Mar 1;5(1):1-8.
Kurdyukov S, Bullock M. DNA methylation analysis: choosing the right method. Biology. 2016 Jan 6;5(1):3.
متیلاسیون DNA یک مکانیسم اپیژنتیکی است که بدون ایجاد تغییر در توالی ژنها، بر بیان آنها تاثیر میگذارد.
جهت کشف تغییرات ناشناخته متیلاسیون در کل ژنوم، میتوان از تکنیکهای با بازده بالا (High-throughput) بهره برد؛ برای بررسی الگوی متیلاسیون DNA در نواحی تنظیمی ژنهای خاص میتوان از روشهایی با اساس تبدیل بیسولفیت، هضم با آنزیم محدودکننده، و غنیسازی مبتنی بر تمایل استفاده کرد.

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
هر گونه سوالی دارید، تیم ما آماده پاسخگویی میباشد.
برای برقراری ارتباط بر روی آیکن مورد نظر کلیک کنید. پشتیبانی 24/7

فرصت رو از دست نده و استاد طراحی پرایمر شو