اسامی برندگان قرعهکشی یلدایی وانیار منتشر شد …

زمان تخمینی مطالعه: 15 دقیقه
Gene Runner (ژن رانر) یک برنامه ویندوزی است که میتواند به شما در انجام آنالیزهای مختلف زیستشناسی مولکولی، مانند ویرایش DNA و توالی پروتئین، آنالیز جایگاههای برش آنزیمهای محدودکننده، تجزیه و تحلیل الیگونوکلئوتیدها، آنالیز توالییابی و هیبریداسیون و… کمک کند.
یکی از ویژگیهای نرمافزار، طراحی و ارزیابی پرایمر برای PCR است. Gene Runner میتواند به شما در طراحی پرایمرهای ایدهآل برای توالی DNA مورد نظرتان کمک کند و همچنین ویژگیها و مشکلات احتمالی آنها را بررسی کند، مانند: پرایمر دایمرها، ساختارهای سنجاقسری، یا عدم تطابقها (mismatch). بهعلاوه، نرمافزار میتواند پرایمرهایی برای کاربردهای خاص طراحی کند، مانند: کلونینگ، جهشزایی (mutagenesis)، ژنوتایپینگ SNP، یا اتصال توالیهای DNA به همدیگر با PCR همپوشان (overlap extension PCR)
کد Readseq نرمافزار ژن رانر را قادر میسازد تا فایل فرمتهای زیر را بخواند و ذخیره کند:
IG/Stanford, GenBank (DNA only), NBRF, EMBL, GCG, DNA Strider, Fitch, Pearson/FastA, Plain/Raw, PIR/CODATA (Protein only)
در لیست زیر هم تعدادی از انواع فایلها را بر اساس پسوند آنها که توسط Gene Runner استفاده میشوند، فهرست کردهایم:
*.RET (فایلهای آنزیم محدودکننده) ، *.SEQ (فایلهای توالی پایگاهداده استاندارد GenBank) ، *.PCR (فایلهای نتایج آنالیز PCR) ، *.ORF (فایلهای قالب خوانش باز یا Open Reading Frame) ، *.CLN (فایلهای آنالیز کلونینگ) ، و *.ALN (فایلهای تراز چندگانه یا multiple alignment)
پیشنهاد: دوره جامع طراحی پرایمر یک فرصت طلایی برای یادگیری حرفهای طراحی پرایمر با نرمافزار Gene Runner و سایر ابزارهای پرکاربرد در این حوزه است.

در این بخش از مقاله، قصد داریم یک مثال طراحی پرایمر را با استفاده از نرمافزار Gene Runner پیش ببریم.
در این مثال، میخواهیم برای دوطرفِ یک پوزیشنِ کروموزومیِ خاص که مربوط به یک واریانت پاتوژنیک در کودکی مبتلا به آلبینیسم است، طراحی پرایمر انجام دهیم. فرض کنید که این واریانت در نتیجه تست توالییابی نسل جدید (NGS) برای بیمار گزارش شدهاست.
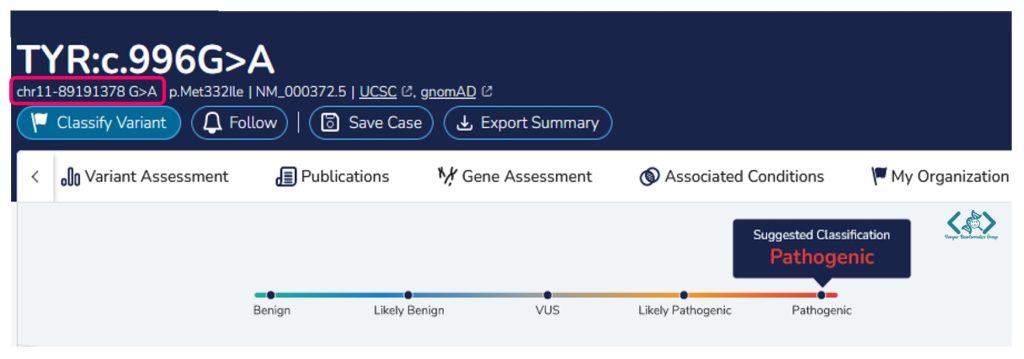
در این مثال، هدف ما چیست: بتوانیم یک محصول PCR داشتیم که پوزیشن واریانت پاتوژنیک بیمار در آن قرار گرفته. بعد از PCR، محصول را برای توالییابی سنگر ارسال میکنیم تا با خوانش توالی بتوانیم وجود یا عدم وجود واریانت را تایید یا رد کنیم.
در این بخش، گام به گام مراحلی که برای طراحی پرایمر طی میکنیم، را برای شما شرح دادهایم.
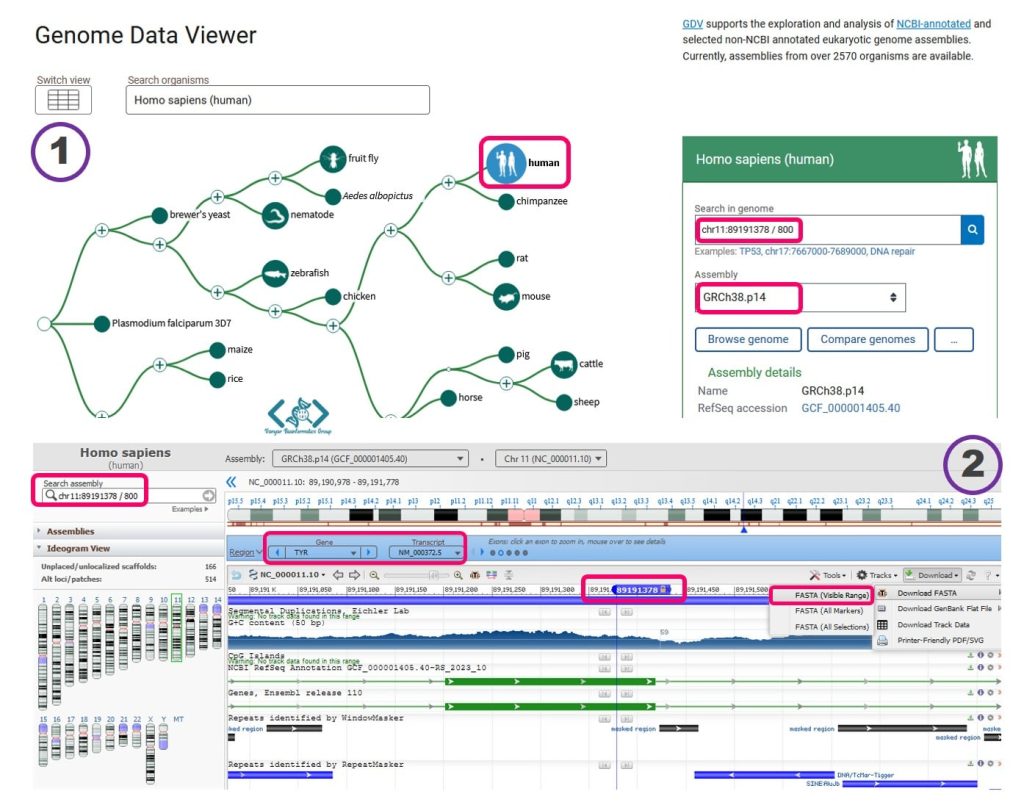
یک راه آسان برای دسترسی به توالی نوکلئوتیدی، استفاده از سایت GDV است. وارد سایت شوید و مانند بخش اولِ تصویر زیر، به ۴۰۰ نوکلئوتید بالاتر و ۴۰۰ نوکلئوتید پایینتر از واریانت موردنظر خود دسترسی داشتهباشید. با کمک تصویر دوم نیز میتوانید توالی این ناحیه را با فرمت FASTA دانلود کنید:
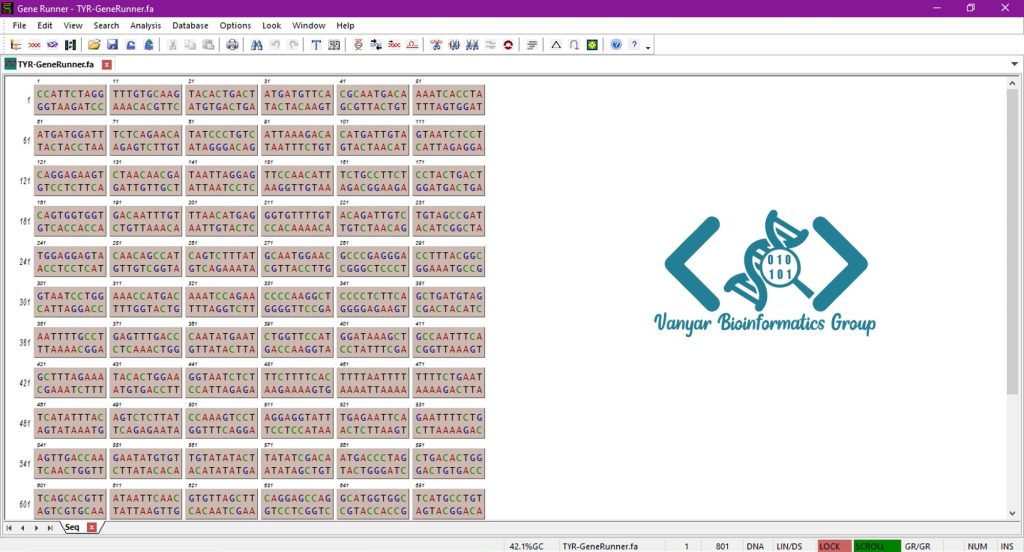
با استفاده از مسیر File > Open در Gene Runner، میتوانید فایل .fa که دانلود کردید را انتخاب کنید و وارد نرمافزار کنید. در اینصورت با چنین صفحهای (پنجره توالی) روبهرو میشوید:
نرمافزار Gene Runner تمام جفت پرایمرهای ممکن را برای منطقه هدف شما طراحی و تجزیه و تحلیل میکند. بهعلاوه، میتواند یک جفت پرایمر را از نظر مناسب بودن برای PCR و نیز ویژگیهای محصول PCR حاصل را آنالیز نماید.
در ادامه مثال، از مسیر Analysis > Nucleic acid > PCR primers به پنجرهای دسترسی پیدا میکنید (تصویر پایین، قسمت A) که در آن پارامترهای مختلفی برای تنظیم کردن محدوده اپتیمم پرایمرهای PCR وجود دارد. در ادامه، همه پارامترها را برای شما توضیح میدهیم تا بتوانید آنها را بهدرستی تنظیم کنید.
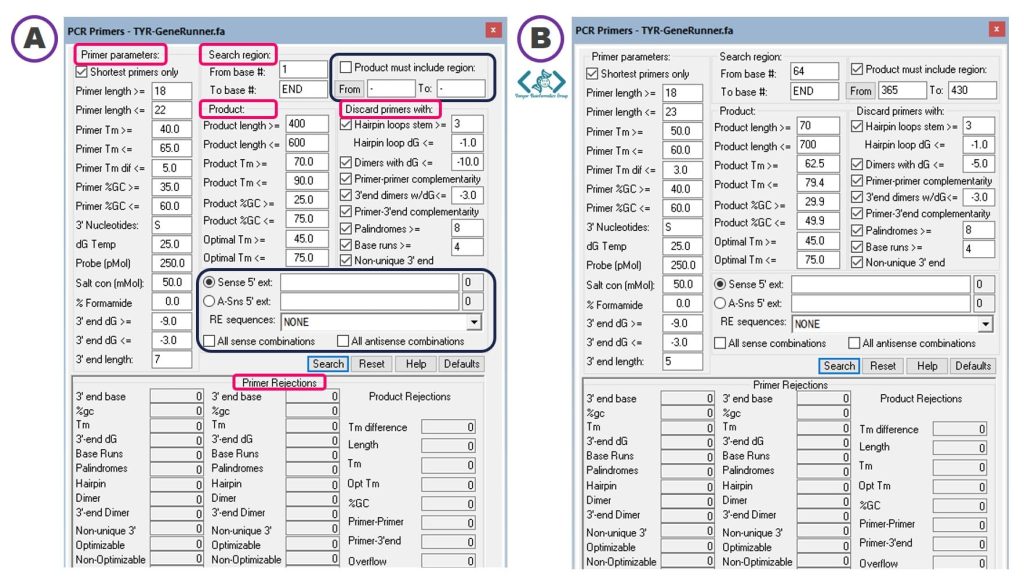
در این بخش، محدودیتهایی مانند طول پرایمر، %GC، Tm و پارامترهای مرتبط مورد نیاز برای انتخاب پرایمر را میتوانید تنظیم کنید. معیارهای دیفالت معمولاً پرایمرهای خوبی به شما ارائه میدهند.
با اینحال میتوانید در مقاله طراحی پرایمر چیست؟ طراحی پرایمر به زبان ساده و کاربردی برای PCR، بیشتر با محدوده ایدهآل پرایمرهای PCR آشنا شوید.
ما معیارهای جستجوی پرایمر را برای این مثال مانند تصویر بالا، قسمت B تنظیم کردهایم.
چک باکس Shortest primers only کوتاهترین پرایمر را که معیارهای موردنظر شما را دارند، انتخاب میکند.
طول پرایمر (Primer length): تعیین آستانه بالا و پایین برای پرایمرها
دمای ذوب پرایمرها (Primer Tm): تعیین حدود نقطه ذوب برای پرایمرها
اختلاف Tm بین پرایمرها (Primer Tm dif): حداکثر اختلاف بین Tmهای یک جفت پرایمر
درصد GC پرایمرها (Primer %GC): تعیین آستانه بالا و پایین برای درصد بازهای G و C در توالی پرایمرها
نوکلئوتیدهای سر 3’ (3’ Nucleotides): این پارامترِ ژن رانر روی S ست شده که یعنی نوکلئوتید C یا G بایستی در انتهای 3’ پرایمر قرار بگیرد. دلیل؟ چون اتصالات C با G قویتر و پایدارتر از A با T است.
دمای دلتا جی (dG Temp): برای محاسبه انرژی آزاد گیبس (dG) مورد نیاز است. این مقدار بر محاسبه همه ΔGها (دلتا جی سر 3’، دلتا جی hairpin loop، و دلتا جی پرایمر-دایمر) اثر میگذارد.
پروب (pMol): لازم برای محاسبه Primer Tm
غلظت نمک (Salt con): مقدار پیشفرض 50 میلی مولار است که غلظت نمک توصیه شده برای اکثر واکنشهای PCR است.
دلتا جی انتهای 3’ (3’-End dG): درواقع ΔG برای 7 باز آخر (پیشفرض) انتهای 3’ است.
در این قسمت از Gene Runner، جستجوی پرایمر را به یک منطقه خاص محدود میکنید.
از نوکلئوتید شمارهی؟ (From base): اولین بازِ ناحیه جستجو است. پیشفرض 200 باز قبل از اولین باز محصول PCR است.
تا نوکلئوتید شمارهی؟ (To base): آخرین باز منطقه جستجو است. پیش فرض 200 باز بعد از آخرین باز محصول PCR است.
در این بخش از ژن رانر میتوانید ویژگیهای محصول PCR مانند طول، درصدGC، و Tm محصولِ تولیدشده توسط پرایمرهای انتخابی را محدود کنید.
طول محصول (Product length): تعیین محدودیتهای بالا و پایین برای طول محصولات PCR
دمای ذوب محصول (Product Tm): تعیین حدود نقطه ذوب محصول. Tm محصول به درصد GC وابسته است و در صورت تغییر پارامترهای درصد GC محصول، بهطور خودکار تنظیم میشود.
درصد GC محصول (Product %GC): تعیین محدودیتهایی برای %GC محصول. در صورت تغییر پارامترهای Product Tm، بهطور خودکار تنظیم میشود.
مقدار Tm بهینه (Optimal Tm): این دما، درواقع دمای اتصال (annealing) است.
این بخش فیلترهای اعمالشده روی پرایمرها را تنظیم میکند. دقیقترین جستجوی پرایمر با فعال بودن همه فیلترها اتفاق میافتد. فیلترها از انتخاب پرایمرهایی که ممکن است پرایمرینگ اشتباه داشتهباشند یا پرایمر-دایمر و غیره تولید کنند، جلوگیری میکند.
گزینه Hairpin loops stem طول قابلقبول ساقه در ساختارهای سنجاقسری را تعیین میکند.
چکباکس hairpin loop dG حداکثر ΔG قابلقبول برای ساختارهای hairpin loop را مشخص میکند.
پارامتر Dimer with dG آستانه پایداری پرایمر برای اتصال به خودش را نشان میدهد.
باکس Primer-primer complementarity، هر جفت پرایمر با قابلیت پرایمر-دایمر را که از حد پایداری تعیین شده توسط پارامتر Dimer with dG فراتر برود، حذف میکند.
گزینه 3′ end dimers w/dG هم دایمرهایی را که انتهای 3’ پرایمر را درگیر میکنند فیلتر میکند.
چکباکس Primer 3′ end complementarity جفتهای پرایمر را برای دایمرهایی که انتهای 3’ هر دو پرایمر را درگیر میکنند و از حد تعیینشده توسط پارامتر 3′ end dimers w/dG فراتر میروند، فیلتر میکند.
گزینه Palindromes پرایمرهایی با توالی پالیندرومیکِ بیشتر از حد مجاز را حذف میکند.
پارامتر Base runs تعداد تکرارهای تکنوکلئوتیدی قابلقبول در توالی پرایمر را درنظر میگیرد.
گزینه Non-unique 3′ end به شما کمک میکند تا مشکلات بالقوه را با پرایمرهایی که انتهای 3’ غیر منحصربهفرد دارند شناسایی کنید، که ممکن است باعث پرایمینگ کاذب شود.
نرمافزار Gene Runner نتایج جستجوی پرایمر PCR را در باکسِ فیلتر PCR نمایش میدهد. در این باکس میتوانید تعداد کل پرایمرها و محصولات پذیرفتهشده و ردشده را ببینید. اگر پرایمرهای مناسبی برایتان پیدا نشده، پارامترهای انتخابی خود را تغییر دهید.
بعد از کلیک روی گزینه search، تمامی پرایمرهایی را که نرمافزار توانسته با معیارهای شما طراحی کند مشاهده میکنید. در جدولی که Gene Runner بهعنوان نتایج ارائه میدهد، اطلاعاتی مانند پوزیشن قرارگیری پرایمرها، طول آنها، Tm ها، درصد GC، توالی نوکلئوتیدی و… وجود دارد.
برای نمایش پرایمرها بر روی توالی و دیدن جایگاه آنها و محصول PCR، کافیست باکس کنار پرایمرها را انتخاب کنید. همچنین برای بررسی دقیقتر خصوصیات پرایمرها، میتوانید مانند تصویر زیر، با کلیکراست روی ردیف پرایمرِ انتخابشده (آبیشده)، گزینه Show Oligo Analysis را بزنید.
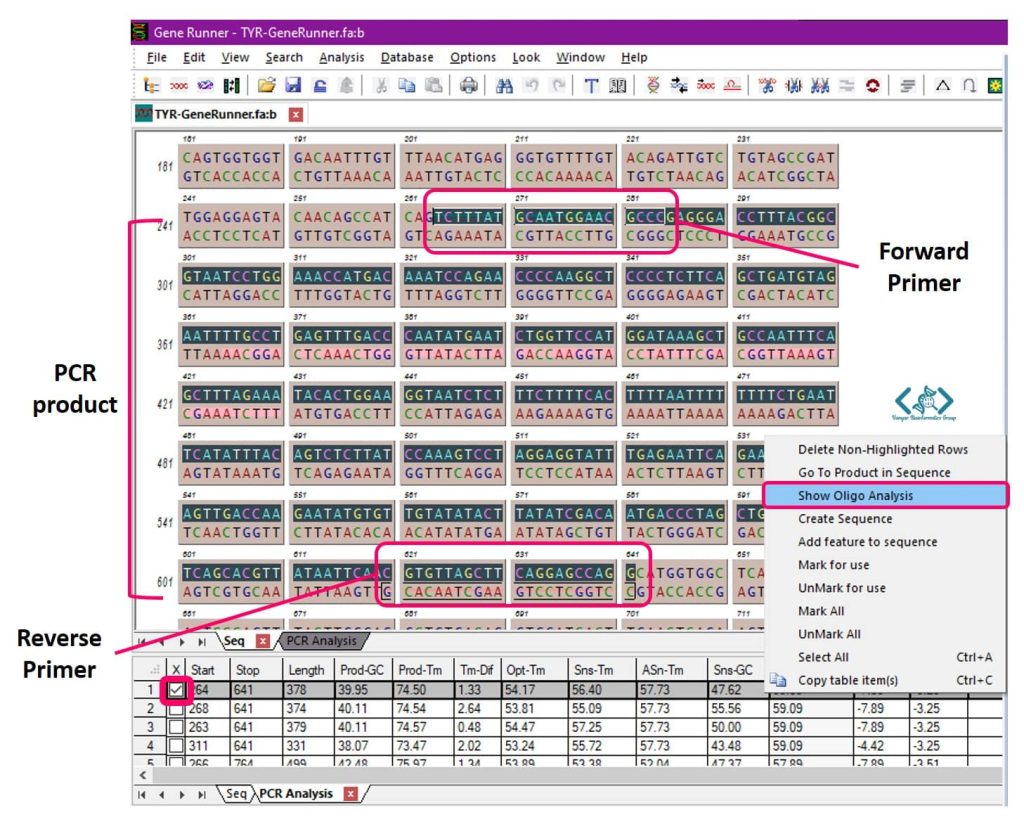
گزینه Oligo Analysis از مسیر Analysis > Oligo در صفحه اصلی نرمافزار ژن رانر هم قابلدسترسی است. پنجره Oligo Analysis به محقق امکان میدهد تا مستقیماً الیگونوکلئوتید مورد نظر خود را وارد کند یا یکی از جفتپرایمرهای جدول را انتخاب کرده و آنالیز کند.
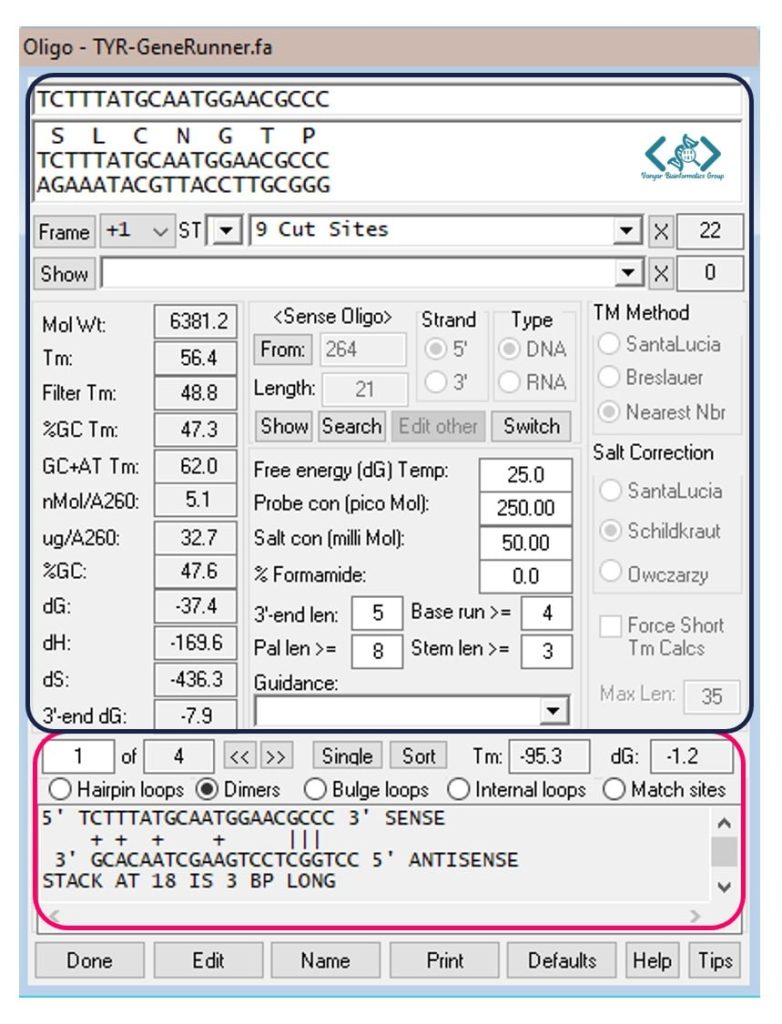
در تصویر بالا و کادر سرمهایرنگ، پارامترهایی مانند توالی پرایمر، جایگاههای برش آنزیمی، وزن مولکولی محاسبهشده برای الیگونوکلئوتید، Tm، درصد GC، انرژی آزاد الیگو (dG)، دلتا جی انتهای 3’، پوزیشن نوکلئوتیدی پرایمر و طول آن، تعداد نوکلئوتیدهایی که بهعنوان سر 3’ پرایمر در نظر گرفتهشده، طول توالی پالیندرومی و… را مشاهده میکنید.
بخش مربوط به ساختارهای ثانویه (سنجاقسر، دایمرها، لوپهای برآمده یا Bulge loops، و لوپهای داخلی یا Internal loops) در پایینِ پنجره (در تصویر بالا، کادر صورتیرنگ) به شما ارائه میشود. لوپهای برآمده و لوپهای داخلی انواعی از دایمرها هستند. Tm در این کادر، دمای ذوب ترمودینامیکی برای ساختارِ نمایش داده شدهاست. Tm برای سنجاقسر (hairpin)، دمایی است که بالاتر از آن سنجاقسر دیگر پایدار نیست. مقادیر Tm در این بخش فقط باید برای مقایسه پایداری نسبی ساختارها استفاده شوند. dG هم انرژی آزاد دایمر یا سنجاقسر است.
در این مرحله، بعد از بررسی خصوصیات پرایمرها، بایستی یک جفت را انتخاب کنید. سپس توالی آنها را (هم فوروارد و هم ریورس) کپی کرده و در جهت 5’ به 3’ در Primer BLAST وارد کنید تا بتوانید اختصاصیت پرایمرها را با مشاهده محصولات PCR بررسی کنید.
پیشنهاد…… اگر علاقهمندید، مقاله طراحی پرایمر در سایت NCBI با ابزار Primer BLAST را بخوانید….
اگر پرایمرهای شما بر اساس NCBI اختصاصی بودند، میتوانید از آنها در PCR خود استفادهکنید. در غیراینصورت، احتمالاً باید جفتپرایمر دیگری را از لیست انتخاب کنید و یاااااا معیارهای طراحی پرایمر را بازبینی کرده و درجه سختگیری را کمی کاهش دهید.
در مثالِ این مقاله، یک جفت پرایمر که Gene Runner به ما پیشنهاد دادهبود را در Primer BLAST بررسی کردیم و نتایج حاکی از آن است که این پرایمرها احتمالاً دو محصول با سایزهای ۳۳۱ و ۳۲۶ جفتباز میتوانند تکثیر کنند. محصول ۳۳۱ نوکلئوتیدی، همان محصول موردنظر ماست (ژن TYR) اما محصول دوم مربوط به ناحیهای از ژنوم است که نمیخواهیم تکثیر شود.
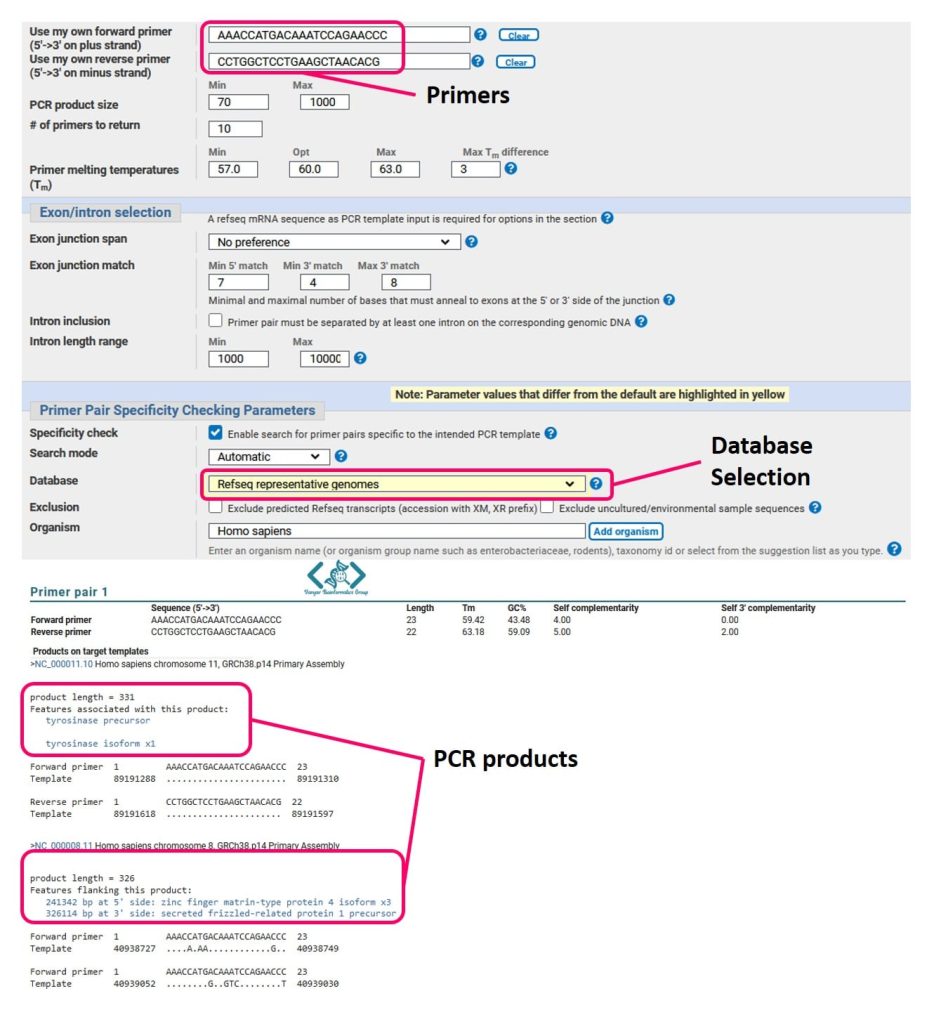
امااااا وقتی به بخش معرفی پرایمرها دقت میکنیم، دو پرایمر Forward داریم BLAST فقط به پیداکردن تشابه اهمیت داده و وجود دو پرایمر فوروارد منطقاً ایجاد اختلال در PCR نمیکند. پس این محصول را میتوانید نادیده بگیرید و اختصاصی بودن پرایمرها را تایید کنید.
در این بخش، فایل نصبی نرمافزار برای ویندوز ۶۴ و ۳۲ بیتی قابل دانلود است. آخرین نسخه این نرمافزار 6.5.52 است که در سال 2019 منتشر شده و به صورت رایگان در دسترس میباشد. این نرمافزار فقط برای سیستمعامل ویندوز موجود میباشد.
در این مقاله از مجله گروه بیوانفورماتیک وانیار، نرمافزار جذاب Gene Runner را به شما معرفی کردیم. همچنین، قدمبهقدم یک مثال طراحی پرایمر را پیش بردیم تا به این وسیله تنظیمات و بخشهای مختلف نرمافزار را به شما نشان دهیم. در انتهای مقاله نیز فایل قابلنصب نرمافزار را قرار دادیم تا برای دانلود ژن رانر از آن استفادهکنید.
آیا تا به حال از Gene Runner برای طراحی پرایمر استفاده کردی؟ خوشحال میشیم چالشها و تجربههاتو با ما به اشتراک بگذاری 😊
Gene Runner یک نرمافزار برای آنالیز و تجزیه و تحلیل توالی DNA است. با استفاده از این نرمافزار میتوان اعمال مختلفی چون مشاهده فایلهای حاصل از توالییابی سنگر، طراحی پرایمر برای PCR، کلونسازی ژنها و … انجام داد.
بله، Gene Runner یک نرمافزار کاملا رایگان میباشد و تمامی امکانات آن به صورت رایگان در دسترس کاربران است.
این نرمافزار فقط برای سیستمعامل ویندوز در دو نسخه 32 و 64 بیتی در دسترس است.
پیشنهاد: افزایش فرصتهای شغلی و تحقیقاتی با شرکت در دوره بیوانفورماتیک عمومی و کسب مهارتهای اساسی بیوانفورماتیک.


تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
هر گونه سوالی دارید، تیم ما آماده پاسخگویی میباشد.
برای برقراری ارتباط بر روی آیکن مورد نظر کلیک کنید. پشتیبانی 24/7

فرصت رو از دست نده و استاد طراحی پرایمر شو