پیش ثبتنام دوره NGS بالینی آغاز شد …

زمان تخمینی مطالعه: 18 دقیقه
Oligo 7 یک برنامه چندعملکردی است که الیگونوکلئوتیدهایی را در یک فایل توالی جستجو و انتخاب میکند تا بهعنوان پرایمر برای PCR، توالی یابی DNA، جهشزایی هدایتشده و کاربردهای هیبریداسیون استفاده شوند.
الیگو ۷ میتواند دماهای هیبریداسیون/ذوب و ساختار ثانویه الیگونوکلئوتیدها را بر اساس نزدیکترین مقادیر ترمودینامیکی مجاور (nearest neighbor thermodynamic values) محاسبه کند.
همچنین محلهای شناسایی آنزیم محدودکننده را در توالی جستجو میکند، فریمهای خواندن باز (ORF) و موارد دیگر را هم تجزیه و تحلیل میکند.
فرمتهای فایل توالی اسیدنوکلئیک که برای خوانش در Oligo 7 قابلقبول هستند، عبارتند از:
Text (ASCII), EMBL, GenBank, Entrez Flat Files
و فایلهای خروجی از اکثر برنامههای تحلیل توالی که قادر به تولید فایلهای متنی سادهاند.
بخش معرفی را با پنجره پیشفرض در نرمافزار الیگو ۷ شروع میکنیم.
هنگامی که یک فایل توالی باز میشود، الیگو ۷ بهطور پیشفرض یک پنجره بهنام “Sequence” را نمایش میدهد:

این پنجره، پنجره اصلی نرمافزار است و شامل عناصر، عملکردها و قابلیتهایی میباشد که در ادامه توضیح میدهیم.
در قسمت بالای پنجره Sequence سه جدول داریم:

در جدول A اطلاعاتی درباره توالی DNA را داریم. بهترتیب شامل:
طول توالی (در اینجا ۵۵۱ نوکلئوتید)
فریم باز خوانش (ORF)
طول الیگونوکلئوتیدی که میخواهیم انتخاب کنیم (در اینجا ۲۲ نوکلئوتید) و پوزیشن شروع آن بر روی توالی (در اینجا از نوکلئوتید شماره ۲۶۸ تا ۲۸۹)
دمای ذوب یا Tm الیگونوکلئوتید انتخابشده (در اینجا 62.2 درجه)
در جدول B اطلاعاتی درباره الیگونوکلئوتیدهایی داریم که بهترتیب بهعنوان پرایمر فوروارد، پرایمر ریورس، الیگوی بالایی و الیگوی پایینی انتخاب میکنیم. این اطلاعات عبارتند از: طول الیگونوکلئوتیدها و پوزیشن شروع آنها بر روی توالی. بهعلاوه در انتهای جدول هم طول محصول PCR را مشاهده میکنید. که در اینجا بهخاطر وجود یک جفت پرایمر خارجی و یک جفت پرایمر داخلی، نرمافزار دو محصول با طول ۱۷۴ و ۸۹ نوکلئوتید را نمایش میدهد.
در جدول C یا Feature تمام ویژگیهای (annotation) توالی را میتوانید ببینید. همچنین میتوانید یک ناحیه (location) را در توالی برای نرمافزار مشخص کنید (مثلاً ناحیه اگزون یک ژن). بعد از آن، نرمافزار میتواند آن ناحیه را با ویژگی یا نامی که به آن دادهاید، در بطن فایل توالی ذخیره کند.
پیشنهاد: دوره جامع طراحی پرایمر یک فرصت طلایی برای یادگیری حرفهای طراحی پرایمر با نرمافزار Oligo 7 و سایر ابزارهای پرکاربرد در این حوزه است.

در میانه صفحه با این پنجره روبهرو هستیم (بخش Zoom-out در Oligo 7):

در بخش Zoom-out، نمای کلی و گرافیکی از توالی خود را در نرمافزار میبینید.
در بالاترین بخش این قسمت، گراف آبیرنگی را مشاهده میکنید که به آن گراف Tm میگوییم. کادر زرد رنگی که بر روی این گراف قرار گرفته، به این معنی است که توالی نوکلئوتیدی این بخش، در قسمت Zoom-in (بخش بعدی) قابل مشاهده است.
در زیر گراف Tm، دهتا میله موازی قرار دارد که هرکدام مختص پارامتر خاصی هستند.
۵ میله بالاتر به جایگاه الیگونوکلئوتیدهای انتخابشده اشاره دارند. بهترتیب: پرایمر فوروارد، الیگوی بالایی، محصول PCR، الیگوی پایینی، و پرایمر ریورس.
۴ میله بعدی به ویژگیهای ساختاری توالی اشاره میکنند. این ویژگیها بهترتیب عبارتند از:
اول: ناحیه تشکیل hairpin loopهای قوی در توالی که برای طراحی پرایمر اصلاً مناسب نیستند.
دوم: جایگاه تشکیل hairpinهای ضعیف، میتوانید در این نواحی پرایمر طراحی کنید ولی حتماً باید مشخصات پرایمر بررسی شود.
سوم: محل نواحی پالیندرومیک که برای طراحی پرایمر مناسب نیستند. (توصیه میکنیم مقاله تکنیک PCR-RFLP، طراحی پرایمر و انتخاب آنزیم محدودکننده را بخوانید)
چهارم: اگر بهدنبال ناحیه خاصی در توالی خود هستید، با جستجوی توالی موردنظر خود (مثلاً ATGTACGTAGCT)، جایگاه آن را بهرنگ زرد در این قسمت میبینید.
پنجم: بخش feature است و تا زمانی که annotation خاصی برای توالی وجود نداشتهباشد یا به توالی annotation اضافه نکنید، بدون رنگ خواهد بود.
در آخر صفحه این بخش را مشاهده میکنیم (بخش Zoom-in در Oligo 7):

بخش Zoom-in، بهمعنای بزرگنمایی توالی است که شامل قسمتهای مختلفی میباشد. شامل:
نوار مقیاس (scale bar) نشاندهنده موقعیت عددی هر نوکلئوتید در توالی است.
توالی پرایمر فوروارد و الیگوی بالایی را بهترتیب با رنگهای زرشکی و صورتی در بالای رشته مثبت (توالی قرمزرنگ)، و الیگوی پایینی و پرایمر ریورس هم بهترتیب با آبی کمرنگ و آبی پررنگ در زیر رشته منفی (توالی آبیرنگ)، میبینیم.
در زیر این موارد، توالی ترجمهشده از رشته DNA/RNA که به نرمافزار دادهاید، قرار دارد.
بهدنبال آن، ساختارهای ثانویه توالی و ویژگیهایی (features) که در بخش zoom-out وجود داشتند را به همان ترتیب میبینیم.
توالی الیگونوکلئوتیدهای انتخابشده، معمولاً با حروف بزرگ و در صورت عدمتطابق (mismatch) با توالی، با حروف کوچک نمایش داده میشوند.
در این قسمت سعی کردهایم با حل یک مثال طراحی پرایمر به روش اتوماتیک، شما را با بخشهای بیشتری از نرمافزار آشنا کنیم.
فرض کنید میخواهید برای بررسی وضعیت واریانت با مشخصات زیر، در دو طرف آن پرایمر طراحی کنید. سپس محصول PCR را برای توالییابی با تکنیک Sanger به شرکت توالییابی ارسال کنید.
ژن: GRHPR
رونوشت: NM_012203
موقعیت کروموزومی: Chr9:37422763:C>T
ژنوم رفرنس: GRCh37.p13
ابتدا باید توالی ژن را در ناحیه موردنظر دانلود کنید. بهاین منظور به سایت Genome Data Viewer (GDV) رفته و مشخصات جستجو را بهاین شکل وارد کنید:

منظور از Chr9:37422763 / 1000 این است که توالی ۵۰۰ نوکلئوتید بالاتر و ۵۰۰ تا پایینتر از پوزیشن Chr9:37422763 را میخواهیم دریافتکنیم.
سپس با این صفحه مواجه میشویم:
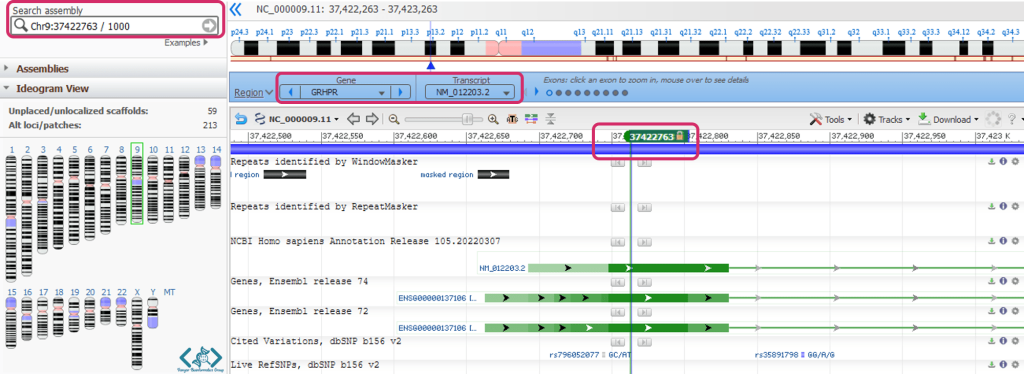
و در مرحله بعد میتوانیم به این شکل توالی را دانلود کنیم:

در این قسمت به طراحی پرایمر با پیشنهاد نرمافزار Oligo 7 یا بهصورت اتوماتیک میپردازیم.
از مسیر File> Open فایلی را که دانلود کردهاید در نرمافزار باز کنید:

همانطور که میبینید طول کل توالی ۱۰۰۱ نوکلئوتید است: ۵۰۰ نوکلئوتید بالاتر و ۵۰۰ تا پایینتر از پوزیشن موردنظر ما.
حالا از مسیر Search > for primers & probes وارد این پنجره میشویم:

گزینه PCR Primers را انتخاب کنید و یکبار وارد آپشن Ranges و یکبار هم Parameters شوید.
با انتخاب Ranges میتوانید تعیین کنید که محدوده طراحی پرایمرها در توالی شما کجا باشد.
مثلاً در اینجا محدوده طراحی را بر روی رشته مثبت (پرایمر فوروارد و الیگوی بالایی) از نوکلئوتید ۱ تا ۴۷۰ و بر روی رشته منفی (پرایمر ریورس و الیگوی پایینی) از ۵۳۰ تا ۱۰۰۱ تعیین کردیم. طول مطلوب محصول PCR هم مشخص کردیم.

بعد با انتخاب Parameters میتوانید پارامترهای طراحی پرایمر را ببینید و تغییر دهید.
گزینه Parameters شامل سه بخش است: General, Constraints, More constraints
پیشنهاد: مقاله آموزش طراحی پرایمر با نرمافزار Gene Runner
پارامترهای بخش عمومی یا General را در این قسمت شرح میدهیم:

Search Stringency این امکان را برای شما فراهم میکند که با یک کلیک، تمام پارامترهای جستجوی پرایمر را با مقادیر ازپیش تعیینشده در Oligo 7 تغییر دهید. با استفاده از این گزینه، میتوانید بدون اطلاع از تنظیمات بهینه طراحی پرایمر، برای الیگونوکلئوتید موردنظر خود تصمیماتی تخصصی خود بگیرید.
در این بخش، شش گزینه سختگیری وجود دارد که بهترتیب عبارتند از:
Very High, High, Moderate, Fair, Low, Very Low
هر درجه سختگیری، پارامترهای طراحی پرایمر را بهشکل خاصی تغییر میدهد.
توجه: با علامت زدن کادر “Automatically change stringency“، در صورتیکه هیچ الیگونوکلئوتید یا جفتپرایمری پیدا نشود، درجه سختی جستجو بهطور خودکار توسط الیگو ۷ کاهش مییابد تا پرایمر طراحی شود.
PCR معکوس (Inverse PCR): وقتی کادر Inverse PCR علامت زده میشود، Oligo 7 پرایمرها را در جهت مخالف انتخاب میکند: پرایمرهای فوروارد در پاییندست، پرایمرهای ریورس در بالادست. (مناسب برای DNA حلقوی)
پارامتر Penalize for Tm / P.E. Discrepancy (جریمه برای اختلاف Tm / P.E): این گزینه امتیازدهی برای انتخاب جفتپرایمر را تغییر میدهد. اگر این گزینه انتخاب شود، اختلافات Tm یا کارایی پرایمر را به حداقل میرساند.
Maximum Number of Selected Primer Pairs: حداکثر تعداد جفتپرایمرهایی که نرمافزار جستجو میکند. معمولاً 3000 جفت (پیشفرض) کافی است.
گزینه Frequency Table:
این جدول فراوانی انواع الیگونوکلئوتیدهای ۶ یا ۷ تایی را در زیرمجموعههای مختلف GenBank فهرست میکند تا از آن برای حذف الیگوهای با تکرار بالا استفاده کند.
بخشهای بعدی هم مربوط به تنظیمات شرایطی هستند که در آزمایشگاه PCR انجام میدهید. تغییر شرایط آزمایش میتواند در Tm پرایمر تغییر ایجاد کند.
تنظیمات این بخش را مانند تصویر قرار دهید.
پیشنهاد: افزایش فرصتهای شغلی و تحقیقاتی با شرکت در دوره بیوانفورماتیک عمومی و کسب مهارتهای اساسی بیوانفورماتیک.

پارامترهای بخش محدودیتها یا Constraints را در تصویر زیر میبینیم:

میتوانید بهترتیبِ باکسهای تصویر، معیارهای طول پرایمر، دلتا جی قابلقبول برای لوپ یا دایمرهای تشکیلشده، پایداری سر 3’ و 5’ الیگونوکلئوتید، پایداری گیره GC، محدوده Tm پرایمرها، و Tm پروب و لوپ را تغییر دهید.
تنظیمات این بخش را مانند تصویر قرار دهید.
پارامترهای بخش محدودیتهای بیشتر یا More Constraints را در این قسمت شرح میدهیم:

گزینه Max Acceptable False Priming Efficiency: الیگو ۷ پرایمرهایی را که بازده پرایمینگ (P.E. #) آنها در محل اشتباه (مکان غیراختصاصی) بزرگتر یا مساوی با مقدار انتخابی (در اینجا ۱۹۰) باشد، حذف میکند.
مقدار بازده پرایمینگ (اشاره به قدرت اتصال پرایمر به الگو) با یک الگوریتم پیچیده محاسبه میشود که دلتا جی دوبلکسها، عدم تطابق، اندازه لوپها و فاصله این عناصر از انتهای 3’ پرایمر را در نظر میگیرد. زمانی که این عدد بالاتر از 220 باشد، احتمال پرایمینگ بیشتر است.
گزینه Min Consensus Priming Efficiency: مربوط به انتخاب پرایمرهای مشترک برای همه فایلهای انتخاب شده
در گزینههای Max Acceptable Homology و Min Consensus Homology نیز میتوانید حداکثر و حداقل مقدار همولوژی پروبها را در محل هیبریداسیون اشتباهشان تعیین کنید.
گزینههای بعدی هم عبارتند از حداکثر تعداد مجاز تکرارهای مونو و دی-نوکلئوتیدی، و حداکثر طول مجاز برای تکرارهای توالی (که باید بیشتر از ۲ باشد)
گزینه Max Degeneracy: توالی پرایمر PCR در صورتی دژنراته نامیده میشود که برخی از پوزیشنهای نوکلئوتیدی آن دارای چندین باز احتمالی باشند. این گزینه پرایمرها و پروبهای حاوی دژنراسیون را فیلتر میکند. تنظیم پیشفرض حداکثر Degeneracy “1” است، که نشان میدهد الیگونوکلئوتید موردبررسی، تنها یک توالی منحصربهفرد دارد.
Template Loop ΔG Threshold و Template Loop Window Size: تنظیمات این بخش بهجهت حذف نواحی با ساختارهای ثانویه قوی در الگوی PCR میباشد.
Max. # of G Bases in a Primer/Probe: در این قسمت میتوانید حداکثر میزان بازهای G در ساختار پرایمر و پروب را تعیین کنید.
تنظیمات این بخش با سختگیری زیاد انتخاب شدهاند. در صورتی که با معیارهای مشخصشده موافقت دارید و یا بعد از تغییر پارامترهای خاص، با کلیک بر روی Apply، تغییرات را ذخیره میکنید.
با کلیک بر روی دکمه search، اجازه میدهید Oligo 7 برای شما طراحی پرایمر کند. سپس پنجره PCR ظاهر میشود که شما با دابلکلیک بر روی هر ردیف میتوانید اطلاعات پرایمرهای طراحیشده را و محصول PCR را ببینید:

در بالای پنجره PCR نمای گرافیکی پرایمرها و محصول PCR را میبینید. سپس جدولی داریم از طول و موقعیت پرایمرها، محصول PCR و مقادیر Tm، درصد GC، بازده پرایمینگ پرایمرها (P.E) و یک امتیاز در ستون آخر.
دقت کنید که وقتی اختلاف Tm محصول PCR و پرایمر بالا باشد، کامنت دریافت میکنید. گفتهمیشود سعی کنید طوری طراحی پرایمر کنید که این اختلاف دما از 25 درجه بیشتر نباشد.
حالا با بستن پنجره PCR، میتوانید با انتخاب هر ردیف از جدول (آبی شدن ردیف)، از منوی Analyze بهترتیب این موارد را برای الیگونوکلئوتیدهای انتخابی یا PCR خود بررسی کنید:

تشکیل دوبلکس (یا همان دایمر)، تشکیل hairpin، ساختار و Tm، جایگاههای پرایمینگ اشتباه، همولوژی، و… . شما در مراحل قبلی برای همه این پارامترها محدوده عددی تعیین کردهاید اما درصورتیکه طراحی دستی با الیگو ۷ انجام داده باشید، میتوانید در این قسمت پرایمرها را بهطور کامل ارزیابی کنید.
درمورد مقادیر مجاز دلتا جی و تعداد مجاز پیوندهای هیدروژنی برای ساختارهای ثانویه پرایمرها میتوانید مقاله طراحی پرایمر چیست را مطالعه کنید.
حالا پرایمرهایی که از طراحی و جایگاه مناسبی برخوردارند را انتخاب میکنیم و بعد از مسیر Analyze> Key info> Selected primers به توالی پرایمرها دسترسی پیدا میکنیم.

نوبت این است که در پرایمر بلاست NCBI (Primer BLAST) اختصاصیت پرایمرها را بررسی کنیم.
توصیه میکنیم مقاله طراحی پرایمر در سایت NCBI با ابزار Primer BLAST را برای آشنایی بیشتر با تنظیمات آن بخوانید.

و اگر مانند اینجا پرایمرهایی اختصاصی نداشتید، پرایمرها را در جایگاههای دیگری انتخاب کنید یا با درجه سختگیری کمتری طراحی کنید:

در بخش زیر نیازهای نرمافزاری و سختافزاری برای نصب برنامه الیگو ۷ بر روی ویندوز و فایل نصبی برنامه را (شامل دستورالعملهای نصب) مشاهده میکنید:
| Windows | |
| نوع کامپیوتر | پنتیوم به بالا |
| سیستمعامل | ویندوز ۹۵ به بالا |
| فضای دیسک و RAM | 20M و 128M |
دانلود نرمافزار برای سیستم عامل ویندوز:
رمز فایل فشرده: www.vanyarbioinf.ir
در این مقاله از مجله گروه بیوانفورماتیک وانیار، نرمافزار پرکاربرد Oligo 7 را به شما معرفی کردیم. بهعلاوه با یک مثال طراحی پرایمر، شما را با تنظیمات و بخشهای مختلف نرمافزار آشنا کردیم. در انتهای مقاله هم فایل قابلنصب الیگو ۷ را قرار دادیم که میتوانید برای برای دانلود نرمافزار از آن استفاده کنید.
آیا شما هم با Oligo 7 پرایمر طراحی کردهاید؟ تجربهها و چالشهای کاری خود را با ما به اشتراک بگذارید…
Primer Design with Oligo Primer Analysis Software v. 7
الیگو ۷ یک نرمافزار چندعملکردی است که در طراحی و آنالیز پرایمرهای توالییابی و PCR، ژنهای مصنوعی و انواع مختلف پروبها از جمله siRNA، چراغهای مولکولی (Molecular beacons)، پروبهای TaqMan و مجموعههای پرایمرهای PCR تودرتو یا Nested PCR کاربرد دارد.
خیر، الیگو ۷ نرمافزاری رایگان نیست و برای دسترسی به بخشهای مختلف نرمافزار باید لایسنس خریداری کنید.
اما در این صفحه، نسخه کامل نرمافزار را میتوانید دانلود کنید.

تیم تولید محتوای گروه بیوانفورماتیک وانیار در تلاش است تا بهترین آموزشهای کوتاه در زمینه بیوانفورماتیک و زیستشناسی را تهیه نماید. صحت محتوای این صفحه توسط کارشناسان گروه بیوانفورماتیک وانیار بررسی شده است.
عضویت در مجله وانیار
جدید ترین مقالات در ایمیل شما!
با عضویت در مجله بیوانفورماتیک وانیار ، برترین مقالات را در ایمیل خود دریافت کنید.
هر گونه سوالی دارید، تیم ما آماده پاسخگویی میباشد.
برای برقراری ارتباط بر روی آیکن مورد نظر کلیک کنید. پشتیبانی 24/7

فرصت رو از دست نده و استاد طراحی پرایمر شو